随着基因组学和分子生物学技术的进步,基于第二代测序的SNP芯片和基因分型测序(GBS)等高通量分子标记技术得到了发展。这些高通量技术平台在各个研究领域都有重要应用,包括在不同遗传物质中发现SNP位点、在作物育种过程中选择遗传背景以及全基因组关联研究。然而,由于其相对较高的成本和复杂的实验程序,将这些高通量技术平台应用于作物的分子标记辅助育种具有挑战性。
在作物分子标记辅助育种的实际应用中,通常会利用与感兴趣的重要农艺性状相关的几个功能多态位点。基于PCR技术的分子标记被设计用于这些位点,促进遗传谱系的辅助选择。
在这种方法中,在Taq DNA连接酶的帮助下,只有当左右寡核苷酸探针完全互补靶DNA序列时,才会发生连接反应,在它们之间没有间隙。如果这两个探针之间有任何间隙,则连接反应会受到阻碍。随后,通过荧光扫描和基于测序峰位移的凝胶定位比对,考虑最后三个未标记的探针碱基差异,确定同一基因座的基因型变化。通过检查最后七个未标记的探针碱基差异,识别出不同的位点,从而能够精确检测SNP位点。
多重PCR扩增:获取靶位点所在的片段。
多重LDR:生产不同长度的检测产品。
测序:通过测序电泳获得不同的峰。
SNP结果和数据分析的解释。
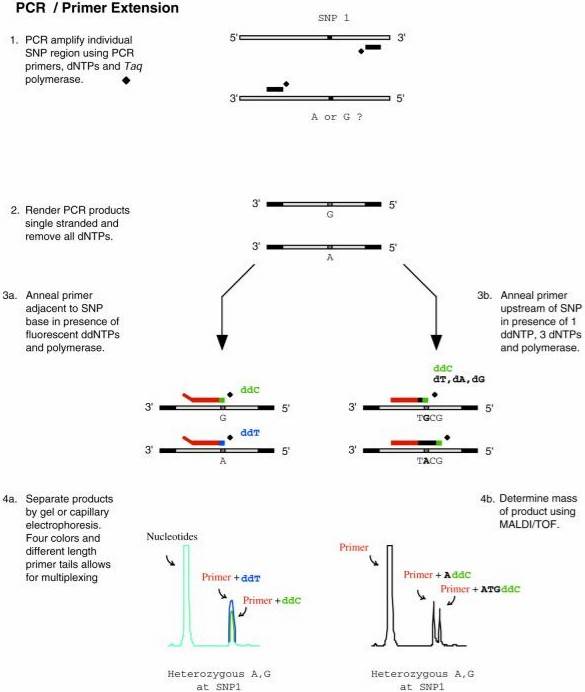
通用性:适用于任何多态位点和各种SNP位点的基因分型,无需引入限制性内切酶位点。
准确打字,检测率高:准确率可超过99.2%。
快速检测:LDR技术最显著的优势是实验时间短。与Taqman、RFLP、SSCP和DHPLC相比,LDR技术可以更容易地控制检测条件,从而使整个实验持续时间仅为几周。
灵活的实验、高检测率和短实验周期。
基于杂交和连接反应的双重保证,确保结果准确。
实验过程中严格的质量控制和10%的重复控制,确保结果的准确性。
适用于中低SNP分型量表,建议使用多达30个SNP位点和少于1000个样本量。
该检测技术适用于各种中低通量SNP检测规模,如QTL定位研究、候选基因或位点关联分析、分子育种等。
参考: