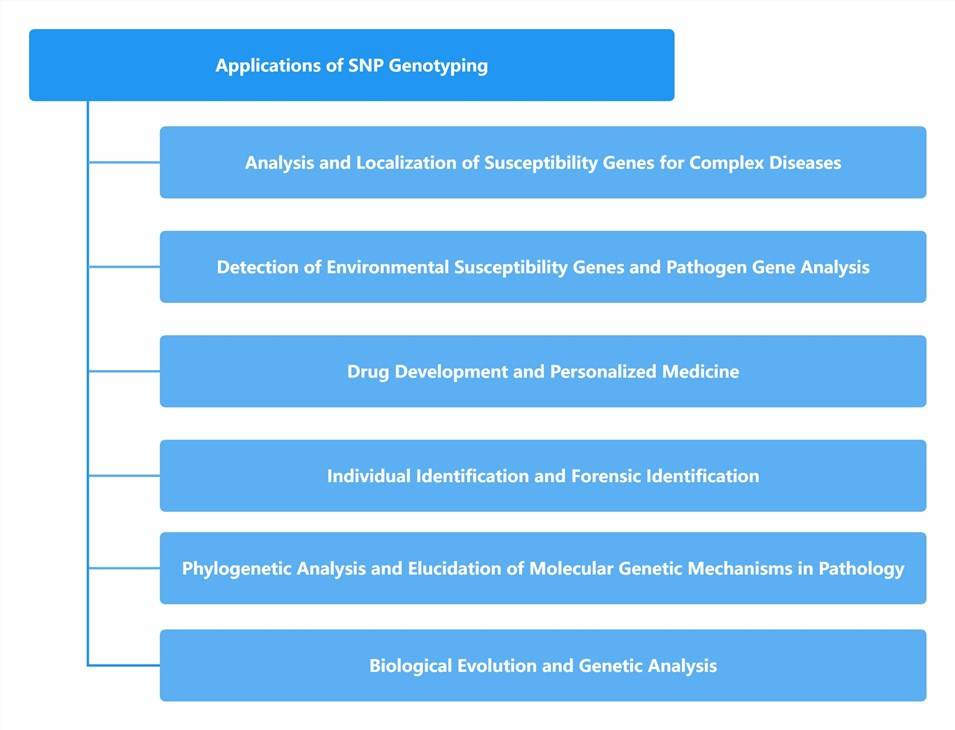
SNP,即单核苷酸多态性(Single Nucleotide Polymorphism)的缩写,指由基因组DNA序列中单个核苷酸的改变(可能是单个碱基的替换、颠换、插入或缺失)引起的遗传多态性。原则上,SNP可以是双等位基因、三等位基因或四等位基因,但在实际应用中,后两者极为罕见,被认为无关紧要。通常,在提及SNP时,重点往往是双等位基因多态性。这些变异可以表现为转换,例如胞嘧啶(C)向胸腺嘧啶(T)的转换,或其互补链上的对应转换,即鸟嘌呤(G)向腺嘌呤(A)的转换。或者,它们也可以表现为颠换,例如胞嘧啶向腺嘌呤(CA)、鸟嘌呤向胸腺嘧啶(GT)、胞嘧啶向鸟嘌呤(CG)或腺嘌呤向胸腺嘧啶(AT)。值得注意的是,转换的发生率始终明显高于其他类别,转换型SNP约占总数的三分之二。
常见的SNP可能出现在基因组的编码区和非编码区。虽然它们在编码区的出现相对不频繁,但它们影响基因功能、从而引起生物性状改变的能力,使其在遗传病研究领域具有至关重要的意义。作为第三代遗传标记,SNP在动物和人类的整个基因组景观中错综复杂地分布。它们与功能基因表现出显著的相关性,拥有极低的突变率,并体现出强大的遗传稳定性。此外,它们适合高通量分析和简化的自动化。值得注意的是,某些SNP位点可能不直接与疾病相关基因的表达相关。然而,由于它们与特定致病基因的邻近性,它们在这些背景下扮演着至关重要的遗传标记角色。
基因芯片具有通量大、准确性高、操作简便等诸多优点,使其特别适用于特定靶位点已预先确定的应用。特别是当靶位点数量超过一定阈值时,基因芯片显示出显著降低的总体费用和更高的效率。
基因芯片技术基于碱基互补杂交原理。它涉及为已知的SNP位点设计探针序列,并用同位素或荧光标记物标记DNA探针。这些标记的DNA探针与目标DNA杂交以获取生物信息。基因芯片利用微阵列技术将数百万个探针集成到显微镜载玻片大小的硅芯片或尼龙膜上。这使得可以同时分析个体的5,000到1,000,000个基因型。
我们的基于微阵列的SNP基因分型服务包括Affymetrix SNP阵列和Illumina SNP阵列。GeneTitan仪器(Axiom阵列板)和GeneChip扫描仪3000 7G系统(盒式阵列)上的Affymetrix基因分型阵列可用于各种情况——通过选择特定的预先设计的基因分型阵列,可用于从靶向到全基因组应用的少量和大量样本。Illumina Infinium或GoldenGate®阵列上的高密度基因分型可实现强大的全基因组关联研究(GWAS),并能准确检测点突变和拷贝数变异。目前,Affymetrix为牲畜和水产养殖物种(水牛、牛、鸡、猪、鲑鱼和鳟鱼)、作物���棉花、玉米、大豆、草莓和小麦)以及生物医学模式生物(人、狗、小鼠和拟南芥)提供基因分型阵列,而Illumina则销售用于人类和非人类物种(牛、狗、玉米、猪和羊)的基因分型BeadArray。由于其高密度、检测准确性、简单的数据分析和研究项目之间的数据交换方便,SNP阵列在某些情况下已被视为首选技术。然而,这些市售的SNP阵列或芯片不能轻易修改以适应个别实验设计。此外,对于没有市售SNP阵列/芯片的物种,无法进行相关研究。
基因芯片的技术基础赋予了以下优势:
高通量:基因芯片能够同时检测大量遗传位点,容量可达1,000,000个位点。
严格的标准化和可重复性:每次实验运行都能在预定位置提供精确的遗传信息,消除了固有的随机性并确保了一致的结果。
卓越的准确性:芯片生产过程贯穿着严格的质量评估,每个检测位点都经过反复审查,从而支撑了结果的卓越精度。
效率:使用基因芯片的实验速度快,最快可在2天内获得结果,有助于研究的快速性。
成本效益:基因芯片提供了一���经济高效的解决方案,特别适合大规模、高通量的基因分析工作。
可定制性:基因芯片可以根据特定的目标位点进行定制,与原位合成的芯片相比,微阵列芯片提供了更强的定制灵活性。
NGS(下一代测序)技术是SNP基因分型的强大工具,因为它们可以高效、准确地发现和基因分型成千上万个SNP,以研究人类、动物和植物的定量、功能和进化基因组学。
NGS是一种从PCR和基因芯片发展而来的DNA测序技术。与Sanger测序相比,NGS测序引入了末端的可逆终止,允许合成和测序同时进行。
在NGS中,单个DNA分子必须被扩增成由相同DNA组成的簇。然后,这些簇被同步复制以增强荧光信号强度,从而便于读取DNA序列。随着读长增加,簇复制的内聚性降低,导致碱基测序质量下降。因此,NGS的读长通常较短,通常不超过500个碱基对。测序后,必须组装来自片段化DNA片段的信息,序列组装过程的错误率在0.1%到15%之间,降低了准确性。
NGS中读长的缩短使得在单次测序运行中能够同时评估数十万甚至数百万个基因序列。这一突破有效地克服了第一代测序方法低通量的限制。单次NGS运行可以熟练地进行多位点基因分型实验,特别是在处理大量样本时,可显著节省时间和降低成本。因此,该技术已在���通量基因测序领域得到广泛采用。
基于下一代测序的单核苷酸多态性(SNP)基因分型有多种方法。
全外显子组测序(WES):WES是一种利用NGS仅对人类基因组中编码蛋白质的外显子区域进行测序的方法。虽然它主要用于研究基因突变与疾病之间的关系,但它也可用于SNP基因分型,因为外显子包含许多SNP位点。
全基因组测序(WGS):WGS涉及对整个基因组的所有区域进行测序,包括编码区和非编码区。该方法可用于检测和基因分型基因组中的所有SNP,提供全面的遗传信息。
靶向测序:靶向测序方法需要对特定感兴趣的基因或SNP位点进行特异性��序,从而获得高度集中的SNP数据。该方法可以根据需要进行定制,以涵盖与特定疾病相关的SNP的选择性靶向,从而能够对与所研究病理相关的遗传变异进行细致的检查。
RNA测序(RNA-Seq):RNA-Seq虽然主要用于研究基因表达谱,但其用途广泛,包括检测单核苷酸多态性(SNP)。这种基于基因转录本审查的多方面方法,有助于在RNA水平上辨别SNP的表达和变异,从而能够全面了解该背景下的遗传多样性和动态。
多重分析:NGS技术通常能够同时对多个样本进行测序,从而提高效率和降低成本。在包括全基因组关联研究(GWAS)在内的大规模单核苷酸多态性(SNP)基因分型项目中,该方法证明了其高度的优势。
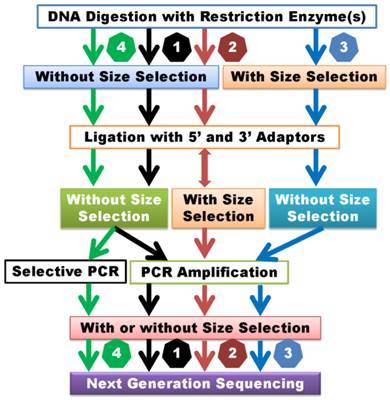
测序分型(GBS):GBS是一种基于第二代测序技术的高通量方法。该方法有助于同时对来自多个个体的DNA样本进行测序并确定其各自的基因型。GBS方案包括文库制备、通过限制性内切酶消化DNA实现的特定基因组区域的靶向测序,以及使用高通量测序仪产生富含单核苷酸多态性(SNP)信息的大量数据集。
dd-RAD(双酶切限制性位点关联DNA测序):dd-RAD是另一种基于第二代测序技术的SNP基因分型技术。该方法需要利用两种不同的限制性内切酶切割DNA,随后对切割产物进行测序,以检测和基因分型SNP位点。
2b RAD:2b-RAD是一种简化基因组代表性测序或限制性位点关联DNA测序策略,旨在以经济高效的方式发现和基因分型遗传变异,适用于具有已知基因组的模型生物和具有未知基因组的非模型生物。该技术定义为通过用限制性内切酶消化DNA来构建文库,并用Illumina测序平台分析后续文库。
PCR在待测位点数量有限的情况下具有高灵敏度、低成本和较短的检测时间(通常在2-4小时内完成)等优点。操作简单。然而,它也有局限性,例如只能检测单个位点��通量低(通常在几个到几百个位点之间),并且无法检测DNA中可能存在的其他突变。可能会出现假阴性和假阳性,因此适用于待测位点数量少、通量要求较低的场景。
连接酶检测反应(LDR)基因分型:该方法主要采用连接酶检测反应(LDR)技术。使用Taq DNA连接酶促进反应,该反应仅在两条短核苷酸探针(每条链各一条)与目标DNA序列完全互补且无任何间隙时发生。通过片段长度的荧光扫描实现检测,从而能够识别SNP位点。
多重SNaPshot SNP基因分型:SNaPshot SNP基因分型是一种同时使用多个PCR对多个已知SNP位点进行遗传分型的方法。它利用DNA测序酶、四种荧光标记的ddNTP、与多态性位点相邻的不同长度的延伸引物和PCR产物模板。引物延伸在单个碱基后终止,在ABI 3730测序仪上进行凝胶电泳后,峰的颜色指示掺入的碱基,根据峰的迁移率确定样品的基因型和与延伸产物对应的SNP位点。
Taqman探针法:该方法涉及在PCR反应体系中加��两种带有不同荧光标记的探针。每种探针与两种等位基因中的一种完全互补。通常,由于探针上5'端荧光基团和3'端淬灭基团的紧密接近,荧光被淬灭。随着PCR的进行,与模板完全互补的探针逐渐被Taq DNA聚合酶的5'→3'外切核酸酶活性切割,导致5'端荧光基团与3'端淬灭基团分离,从而解除荧光淬灭。相反,代表另一种等位基因的探针与模板不完全匹配,因此不会被有效切割,也检测不到荧光。通过使用相应仪器测量荧光值的变化来实现SNP位点检测。
PCR-ARMS(扩增阻碍突变系统):ARMS是一种针对特定SNP设计的PCR扩增方法。通过引物设计,它选择性地扩增包含或不包含特定SNP的DNA片段。
基于PCR的技术为SNP基因分型提供了一系列选择,每种技术都以其独特的特点和对特定研究背景的适应性而著称。
MassARRAY SNP基因分型:基质辅助激光解吸/电离飞行时间质谱(MALDI-TOF MS)是该方法的基础技术。最初,采用PCR扩增包含SNP的基因片段。随后,使用序列特异性引物进行单碱基延伸。���后,样品分析物与芯片基质共结晶,并在真空管内被纳秒(10-9s)激光脉冲激发。该过程导致核酸分子解吸,将其转化为单电荷离子。由于离子质量与电场中离子飞行时间成反比,通过测量真空管内核酸分子的飞行时间来确定样品分析物的精确分子量。因此,可以可靠地检测SNP位点信息。该方法非常适合中高通量SNP分析,特别是在处理十个以上SNP位点时。
CD Genomics致力于以经济实惠的方式提供全基因组范围内的SNP基因分型。我们经验丰富的科学主页可以为您的项目提供专业支持,以满足您的研究目标和预算。